聊聊基因编辑技术的前世今生:ZFN、TALEN、CRISPR

前言
在生命科学的探索之旅中,基因编辑技术一直扮演着至关重要的角色。从最初的锌指核酸酶(ZFN)到如今的Prime Editing技术,基因编辑技术经历了从概念到实践、从粗糙到精细的跨越式发展,为我们理解和操控生命的本质提供了强大的工具。
基因突变
说到基因编辑,我们要先从基因突变说起。基因点突变(point mutation)指的是只有一个核苷酸对发生改变的单基因突变,也称为单碱基替换(single base substitution)。这种突变可以是转换(transition,嘌呤被另一种嘌呤取代,或嘧啶被另一种嘧啶取代)或颠换(transversion,嘌呤和嘧啶之间的替换)。点突变可能是自发的,也可能是由于诱变剂(如某些辐射和化学药剂)引发的。它们可以出现在编码区或非编码区,并可能直接影响蛋白质的功能或基因表达的调控,产生同义突变、错义突变、无义突变或终止密码突变。在编码区,点突变可能导致氨基酸替换、插入或缺失,进而影响蛋白质的结构和功能。在非编码区,点突变可能影响基因表达的调控序列,如启动子或增强子,从而改变基因的表达水平。
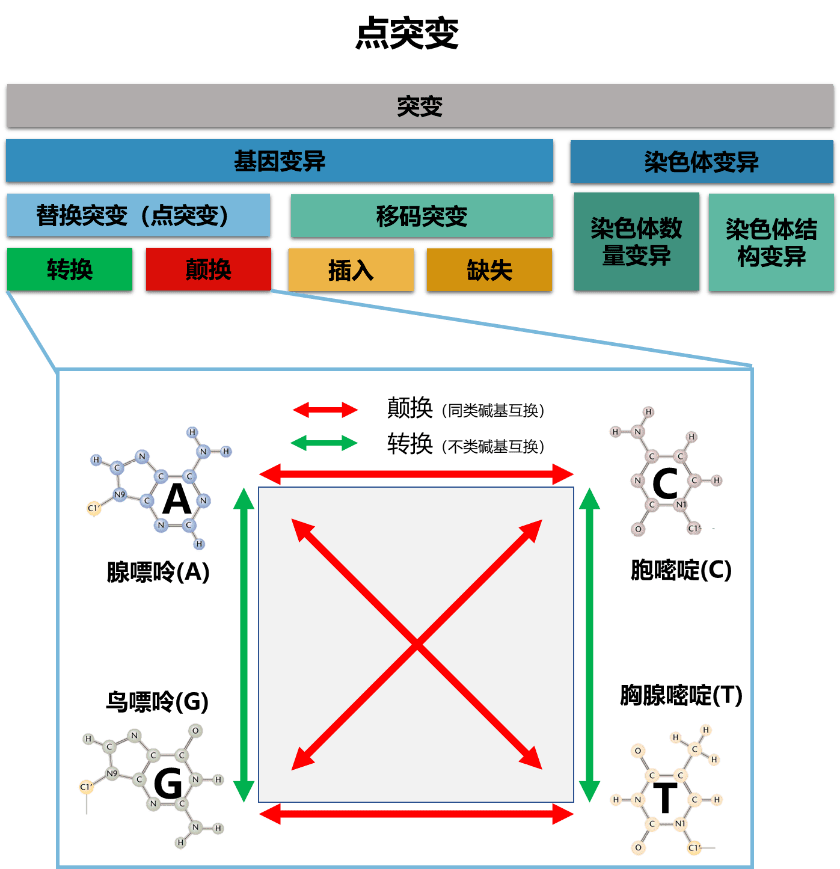
人类有超过75000种疾病和遗传变异相关,其中单碱基突变占的比例最大,我们设想一下,如果我们把突变基因修复成正常基因了,以后患者是不是就可以免于罕见病的困扰了?因此为了这一目标,人们在不断研究更精确的基因编辑工具,通过对特定基因进行精确的编辑,如碱基替换或基因敲除,研究基因功能以及其在生物体中的作用。通过改变基因的特定碱基影响对应的氨基酸序列和蛋白质结构,研究蛋白质结构和功能之间的关系。通过研究药物与靶点的相互作用,为新药研发提供有力支持,也可以用于精确修复致病基因,为治疗遗传性疾病提供新的手段。
那么下面我们来回顾一下基因编辑技术精彩的发展历程。
基因编辑技术的发展历史
一、“指哪打哪的黄金手指”-------ZFN
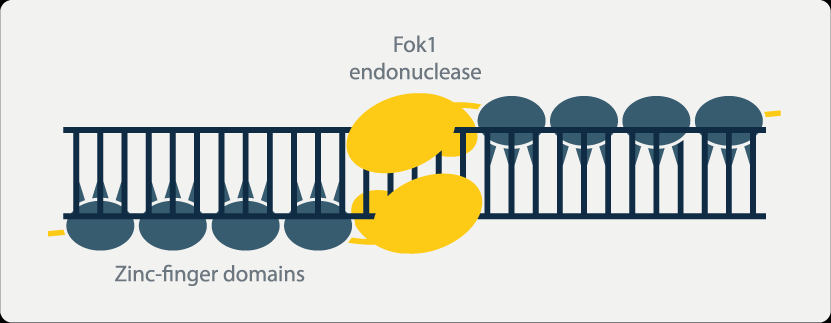
二十世纪九十年代末,Aeron Klug实验室在非洲爪蟾的转录因子中发现锌指蛋白,经人工改造并连接上核酸内切酶后,发展为基因工程编辑工具:锌指核酸酶技术(ZFN)。
锌指核酸酶技术依赖于锌脂蛋白行驶功能,主要用于定点编辑和基因组修饰。锌指蛋白是一类具有DNA结合能力的蛋白质,蛋白内含有锌离子结合的结构域:锌指。每个锌指通常能识别并与3到4个相邻的核苷酸碱基序列结合,通过组合不同的锌指,可以根据需要精确地设计出能够特异性结合目标DNA序列的锌指蛋白。此外,非特异性核酸内切酶Fokl与锌脂蛋白相连后使其具有DNA切割能力,多个锌脂蛋白串联可形成锌脂蛋白组,锌脂蛋白间相互作用产生酶切功能,形成双链断裂,刺激细胞启动自然DNA修复过程,诱发位点特异性重组,达到基因编辑的目的。
但锌指核酸酶技术也存在一定的局限性,如锌指中各个锌指蛋白可以相互作用,影响识别和结合特定核苷酸序列,导致在多个锌脂蛋白串联成组时可能会错误地切割到两个目的片段之间的片段,该上下文依赖效应将可能导致潜在的脱靶效应。锌指酶识别的目标序列长度是一定的,一些小基因和同源性高的基因可能无法被锌指核酸酶技术有效敲除,大片段基因也难以通过锌指酶技术敲入。此外,锌指核酸酶的装配过程非模块化,锌脂蛋白合成时间长,在大肠杆菌中组装困难,成本高、专利壁垒等一系列问题限制锌指核酸酶技术广泛应用。
2017年11月,第一个评估基于锌指核酸酶(ZFN)的体内基因组编辑治疗(ZFN体内编辑治疗)的安全性和耐受性的人体临床研究开始对第一批受试者给药。这三项首次在人类中进行的非盲递增单剂量1/2期研究主要是为了评估ZFN体内编辑疗法在黏多糖贮积症I (MPS I) (n = 3)、MPS II (n = 9)和血友病B (n = 1)中的安全性和耐受性。三项研究共纳入13名受试者:9名受试者参与MPS II研究,3名受试者参与MPS I研究,1名受试者参与血友病B研究。通过检测到两个MPS II受试者肝脏中白蛋白-IDS融合mRNA和一个MPS I受试者肝脏中白蛋白-IDUA融合mRNA,证明ZFN在体内基因组编辑成功。在1e13 vg/kg剂量下,MPS I受试者10的白细胞艾杜糖苷酶活性短暂升高至正常水平的一半,并有体内基因组编辑的证据。

二、“神话核酸酶”--------TALEN
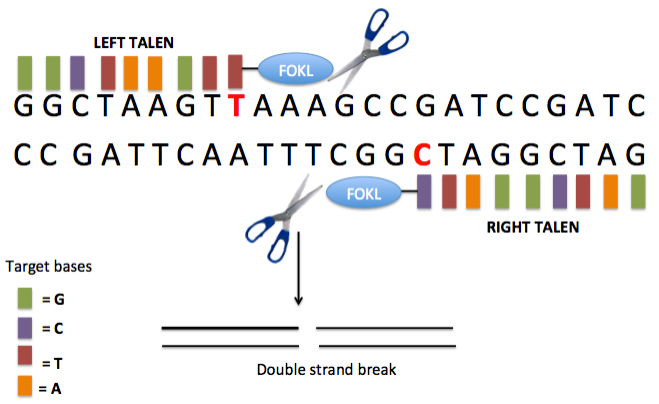
2007年,德国科学家Ulla Bonas在植物黄单胞菌中发现一套精密的微型注射系统,黄单胞菌通过注射一种名为AvrBs3的蛋白质进入植物细胞,伪装成植物的转录因子,为黄单胞菌合成一些黄单胞菌急需的蛋白质,满足其生存和繁衍的需求。往后发现不同的细菌都具有不同的类似的蛋白,该类蛋白被统一称为转录激活因子样效应物TALE。
TALEN是DNA识别结合区域TALE和非特异性核酸内切酶Fokl组合后的产物,通过二聚体化使目的片段产生双链断裂,再激活细胞内的NHEJ修复机制修复DNA双链损伤,在修复的过程中引入indel,达到基因点突变的目的。TALEN比ZFN有更高的特异性,能高效地实现基因组的编辑和修饰,并且TALEN的DNA结合是模块化的,可以方便地组合和修改以适应不同的目标DNA序列,可以灵活地应用于多种生物体,但模块化设计难度较高,需要一定的专业知识和技能。此外,TALEN具有一定的细胞毒性,对细胞生长和分裂产生不利影响。“神话核酸酶”TALEN的出现,悄悄打开了基因编程时代的大门。
2022年11月,基因编辑公司Cellectis公布了其基于TALEN基因编辑的CAR-T细胞的临床前数据,美国FDA已经批准在研产品UCART20x22的IND申请,以启动用于治疗复发性或难治性非霍奇金淋巴瘤 (r/r NHL) 的1/2a期临床试验NatHaLi-01。UCART20x22利用TALEN技术敲除了TRAC基因和CD52基因,降低移植物抗宿主病风险,允许在患者的预处理方案中使用CD52单抗,以增强CAR-T的移植、扩展性和持久性。
三、基因编程时代来临----CRISPR-Cas
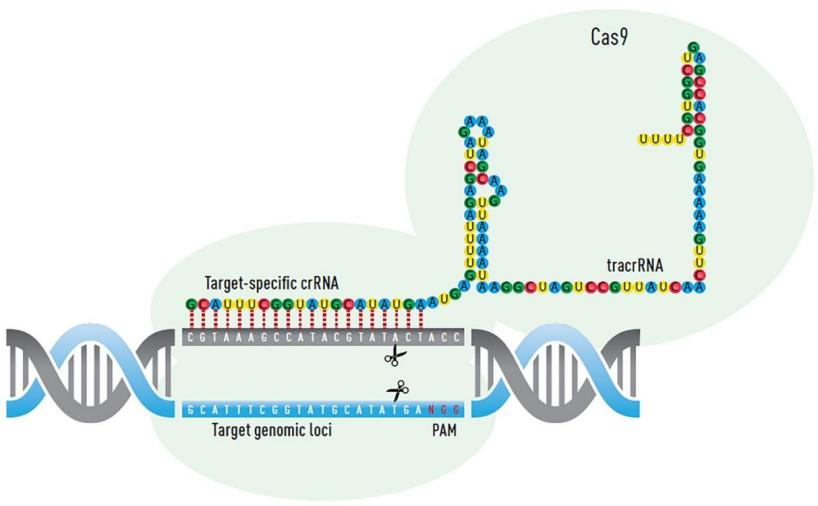
1987年,日本科学家石野良纯在大肠杆菌研究中偶然发现一些每两段相同的序列夹着的一段看起来杂乱无序的序列,1993年,西班牙科学家Francisco Mojica在20种不同微生物中都发现了这种重复结构。2011年,Doudna和Charpentier通过CRISPR序列往后数年间,对这些序列的研究愈加火爆,Doudna和Charpentier凭借相关研究一举拿下了2020年的诺贝尔化学奖。这些神奇的重复序列被称为成簇规律间隔的短回文重复序列CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)。
CRISPR系统是由single-guide RNA介导的核酸酶系统,通过特异的CRISPR序列与靶序列进行碱基配对从而引导Cas9蛋白至特定的切割位点进行切割,通过细胞内的修复机制HDR或NHEJ进行修复,HDR修复精确度高,但修复效率低且有限,细胞还是以NHEJ修复为主。NHEJ修复可能会产生小片段的插入、缺失和替换,实现基因编辑。
CRISPR技术可以精确地识别和切割目标DNA序列,具有很高的特异性,能够在短时间内实现大量细胞的基因编辑,可以应用于多种生物体,与上述两种编辑技术相比,CRISPR技术的优势非常明显,操作简单,效率更高,且成本相对更低。尽管CRISPR技术具有很高的特异性,但仍存在脱靶效应,造成基因组DNA大片段的缺失、易位和破裂,且进行体内实验时Cas9蛋白可能引发免疫反应,影响基因编辑效率。
2024年5月,美国麻省眼耳医院和俄勒冈健康与科学大学联合开展EDIT-101实验性基因编辑治疗。在这项临床试验中,共有14名参与者。其中低剂量(2名参与者)、中剂量(5名参与者)、高剂量(5名参与者)注射给12名17至63岁的成年人,并在2名9岁和14岁的儿童中注射了中剂量,参与者均为都患有CEP290基因突变引起的LCA10。在实验过程中,参与者的一只眼睛接受EDIT-101的单次注射治疗,且过程中未发生与治疗或程序相关的严重不良事件,也没有剂量限制性毒性作用。采用FST评估,6名参与者的视锥体感光细胞有显著改善,其中5名至少在一项其他关键次要结果上有改善,9名参与者(64%)在最佳矫正视力、FST测量的红光敏感性或活动能力测试得分方面有显著改善,6名参与者的视力相关生活质量评分有显著改善。
四、开启精准编辑时代--Base editor
2016年,DavidR. Liu实验室在《Nature》上发表名为“Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[1]”的论文,开发了基于CRISPR/Cas9的单碱基编辑器Base editor,提高了CRISPR系统的特异性和编辑效率。
Base editor系统使用没有酶切活性的dCas9和胞嘧啶脱氨酶融合蛋白,胞嘧啶脱氨酶可结合到基因组DNA形成的R-loop区的ssDNA处,将该ssDNA上一定范围内的胞嘧啶(C)脱氨变成尿嘧啶(U),进而通过DNA复制或修复将U转变为胸腺嘧啶(T),最终实现CG碱基对直接替换为TA碱基对,将基因组上特定C碱基改变为T碱基的技术被称为CBE技术。在这篇文章中,David Liu团队在CRISPR-Cas9的基础上对脱氨酶种类、融合位置及linker进行优化,得到第一代工具rAPOBEC1-XTEN-Cas9,即BE1。为了进一步提高编辑效率、减少indel出现频率,David Liu团队在Cas9 C端融合UGI(Uracil DNA glycosylase inhibitor),得到第二代工具rAPOBEC1-XTEN-Cas9-UGI,即BE2。David Liu团队为了使工具突破50%编辑效率的理论限制,利用具有单链切割活性的nCas9(nickase Cas9)替换dCas9,得到第三代工具rAPOBEC1-XTEN-nCas9-UGI,即BE3。
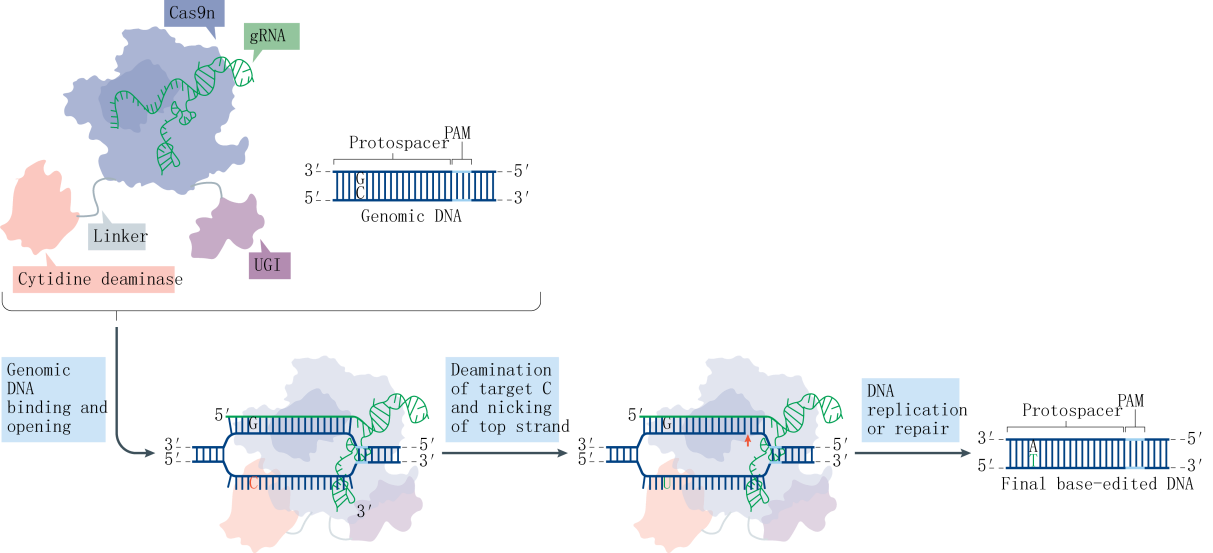
研究显示大部分单碱基遗传病是由G-A突变导致的,有了CBE系统后,下一步亟待解决的便是ABE系统的开发。ABE开发工作的难点在于,自然界并不存在一种DNA腺嘌呤脱氨酶,为解决这一难题,David Liu团队挑选出大肠杆菌来源TadA蛋白作为底物,设计出一套抗生素筛选系统进行定向进化,经过七轮进化,团队筛选得到人工定向进化的腺嘌呤脱氨酶。nCas9与腺嘌呤脱氨酶融合蛋白在sgRNA的引导下靶向基因组DNA时,腺嘌呤脱氨酶可结合到ssDNA上,将一定范围内的腺嘌呤(A)脱氨变成肌苷(I),I在DNA水平会被当做G进行读码与复制,最终实现AT碱基对直接替换为GC碱基对。

进一步研究统计发现,CBE造成indel的概率明显大于ABE,分析后发现细胞内存在UNG(uracil N-glycosylase),当CBE将C碱基脱氨变成U碱基后,UNG识别并切除基因组上的U碱基,造成无碱基位点,形成indel。为了解决这一问题,David Liu团队优化BE3 linker并增加UGI融合个数,得到第四代工具rAPOBEC1-nCas9-UGI-UGI,即BE4,往后进一步对BE4进行密码子优化并添加NLS(Nuclear Localization Sequence)进一步提升工具的效率,得到BE4max。近期,一个团队提出另一条解决道路,开发出GBE系统。GBE是在CBE的基础上引入了外源的UGI(uracilglycosylaseinhibitor)抑制UNG的活性,保护U碱基,提高C到U再到T的转换效率。
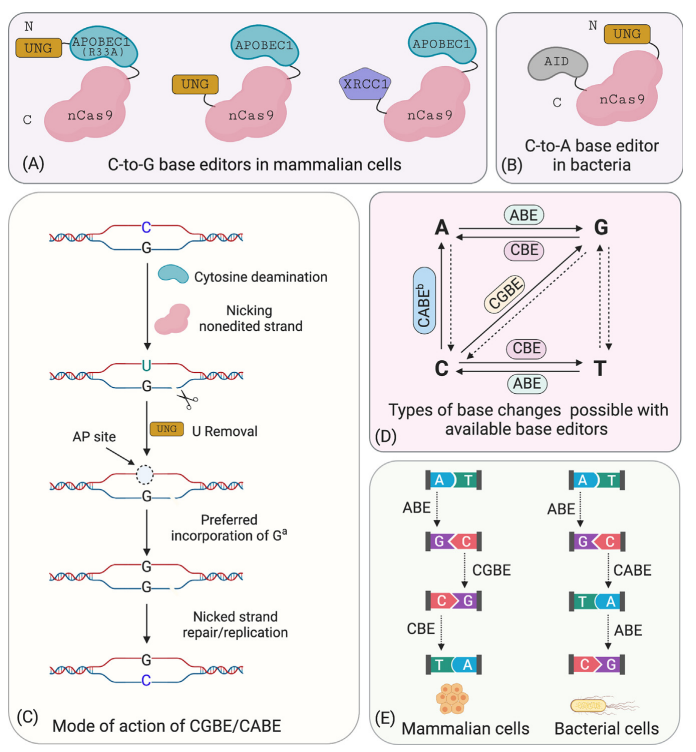
CBE和ABE组合使用可以有效地进行4种碱基转换(C→T, G→A, A→G, T→C),然而除了这4种碱基转换,无法实现另外8种碱基转换(C→A, C→G, G→C, G→T, A→C, A→T, T→A, T→G)以及碱基的插入和缺失,且由于相应的融合蛋白作用区域有一定的范围,因此目标碱基附近的相同碱基可能发生碱基转换,从而降低特异性编辑效率。
2024年1月,广西医科大学第一附属医院开展的针对重型β-地中海贫血症的碱基编辑药物CS-101的研究者发起的临床试验(IIT)研究成功治愈首位患者,达到持续摆脱输血依赖超过两个月,治疗后8周,患者的胎儿血红蛋白浓度上升至~95 g/L,比例上升至~81%,表达胎儿血红蛋白的F细胞比例上升至~80%。CS-101通过采集患者自体造血干细胞,利用高精准变形式碱基编辑器tBE(transformer Base Editor)对患者自体造血干细胞中的HBG1/2启动子区域进行精准碱基编辑,模拟健康人群中天然存在的有益碱基突变,重新激活γ-珠蛋白的表达,重建血红蛋白的携氧功能,再将编辑后的造血干细胞回输至患者体内,使得患者自身血红蛋白浓度达到健康人水平,从而彻底摆脱输血依赖。
五、精准编辑里程碑---Prime editing
2019年,David Liu团队在Nature期刊刊发表名为“Search-and-replace genome editing without double-strand breaks or donor DNA[2]”的论文,标志着PE工具的诞生。
Prime editor复合物通过pegRNA引导下识别PAM并结合与guide RNA互补的目标DNA链,Cas9酶切割PAM上游原始间隔序列第3和第4个核苷酸之间产生一个切口;断裂的DNA单链与PBS结合,逆转录酶生效,沿着PBS进行逆转录,生成编辑序列;编辑序列与目标DNA原序列会竞争性与另一条链通过碱基互补配对结合,未结合的序列则会翘起来,最后被切除。竞争性结合最后会产生两种结果:一种是目标DNA原序列留下,编辑序列被切除,此时目标DNA未被成功编辑,可能会再次被复合物再次识别;另一种是编辑序列留下,目标DNA原序列被切除,此时由于编辑后序列与另一条链序列不匹配,编辑位点形成突起,细胞内DNA错配修复机制进行修复。而修复的结果也分为两种,分别是编辑位点被修改,回归目标DNA最初始的状态;以及另一条链被修复,生产编辑后的DNA双链,成功达到精准基因编辑的目的。
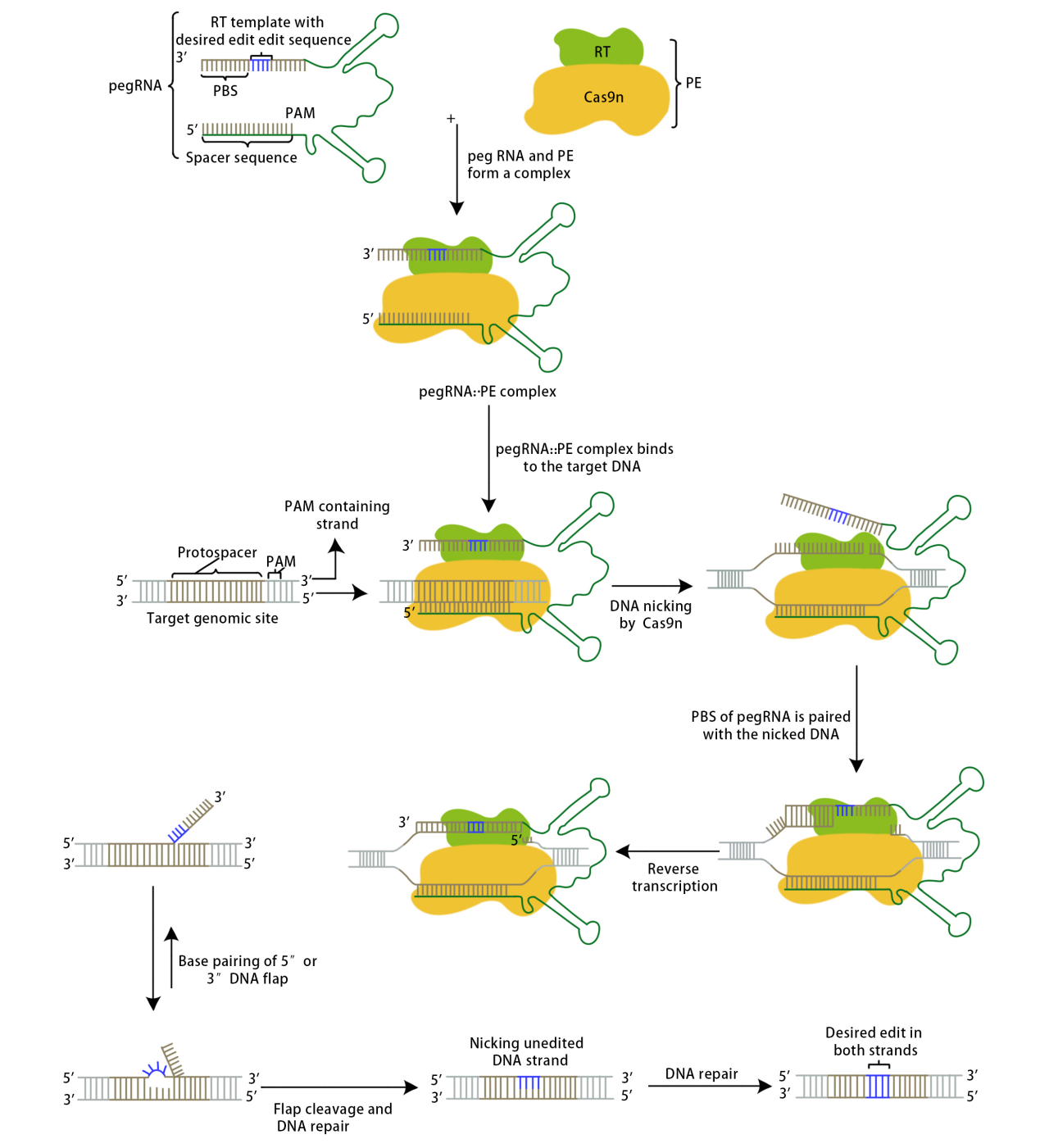
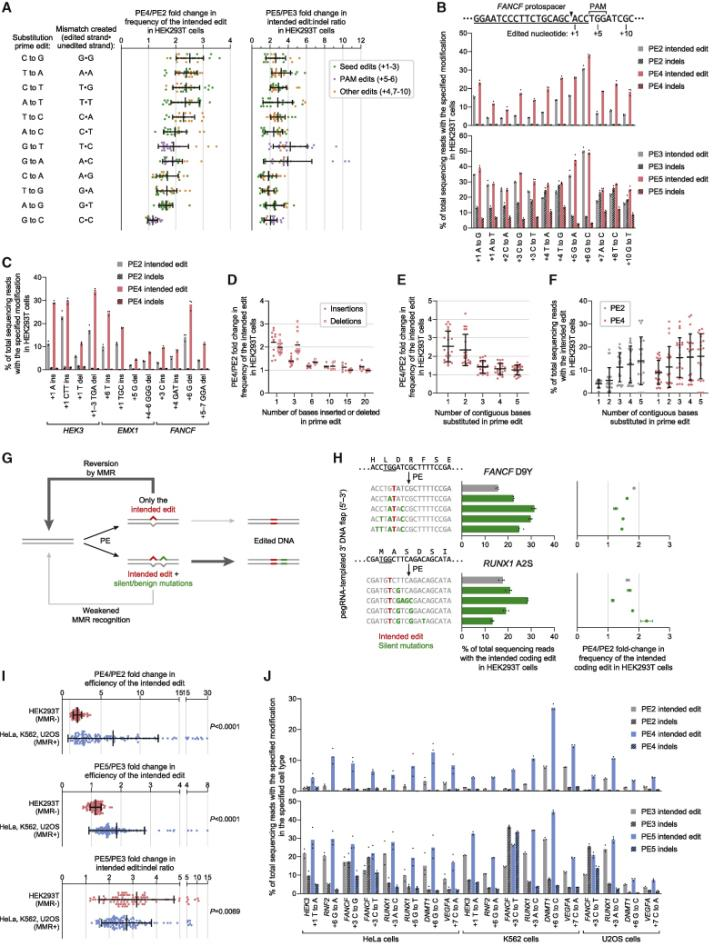
David Liu团队分别进行了PE1、PE2和PE3相关实验。PE1系统使用了一个野生型的M-MLV逆转录酶,通过柔性连接体与Cas9 H840A切口酶的任一末端融合,并与一个pegRNA共转染到HEK293T细胞中。最初的尝试没有检测到可编辑性,后来通过将pegRNA中的引物结合位点(PBS)扩展到8-15个碱基,使PE1能够在HEK3位点目标位点上检测到可转换的点突变。为了提高DNA合成的效率,研究者们对PE1中的RT进行了工程化改造,构建了19种PE1变体,并评估了它们在人类细胞中的编辑效率。引入特定的M-MLV RT突变(D200N+L603W+T330P,称为M3)后,与PE1相比,在五个基因组位点上平均转换和插入编辑效率提高了6.8倍。进一步添加T306K和W313F突变,形成了PE2,它在六个转换或插入编辑中对五个基因组位点的编辑效率比PE1提高了1.3到3.0倍。PE3系统通过使用Cas9 H840A切口酶在非编辑链上制造切口来增强编辑效率,在五个基因组位点上测试时,与PE2相比,编辑效率提高了1.5到4.2倍,最高可达55%。进一步实验发现切口酶往往在DNA双链都产生切口,诱发DNA双链修复机制,引入indel。为此重新设计新加入的sgRNA,使得只有在PAM链编辑之后才在非编辑链上制造切口,PE3b系统平均降低了13倍的indel形成(0.74%),而编辑效率没有明显下降。
2021年,David Liu与普林斯顿Adamson团队合作,开发出PE4、PE5和PEmax系统,并发表名为:Enhanced prime editing systems by manipulating cellular determinants of editing outcomes[3]的文章。他们使用CRISPRi筛选发现DNA错配修复(MMR)途径抑制了Prime editing的效率和精确性。敲低MMR复合体的组成部分(如MSH2、MSH6、MLH1和PMS2)后观察到Prime editing效率的提升。PE4和PE5是在PE2和PE3的基础上通过共表达工程化的MMR抑制蛋白(MLH1dn)使其失去核酸内切酶的功能,瞬时抑制错配修复酶的活性。在六种MMR正常的细胞类型中,包括诱导性多能干细胞(iPSCs)和原代T细胞,PE4和PE5系统相较于PE2和PE3系统,平均提高了7.7倍和2.0倍的编辑效率,同时将编辑/indel比率提高了3.4倍。通过优化逆转录酶密码子、在SpCas9中加入一些氨基酸突变以提升酶切效率、在整个酶两端加入NLS肽段以增加复合酶对核酸的吸附力以及nCas9和逆转录酶之间的肽链连接长度和组成等,开发了PEmax架构,该架构在HeLa细胞中平均提高了2.5倍的编辑效率,在HEK293T细胞中提高了1.2倍。使用PE4max和PE5max系统在与镰状细胞贫血症、朊蛋白病、CDKL5缺乏症、HIV感染和T细胞转移疗法相关的六个基因组位点上进行编辑,观察到显著的编辑效率提升和indel副产品的减少。
2023年8月,David Liu团队在Cell上发表名为:Phage-assisted evolution and protein engineering yield compact, efficient prime editors[4]的文章,开发出新的PE6及其变体PE6a-g。利用噬菌体辅助进化技术(PACE),在数百小时的时间里,携带原始PE编辑器的噬菌体经历了数千代的进化,迅速产生具有全新结构的逆转录酶,生成了比当前PEmax编辑器更小、效率更高的PE6。通过AAV递送系统在小鼠体内测试了PE6,能够在小鼠大脑皮层中实现高达40%的loxP序列插入,比以前的PE编辑器提高了24倍。此外,David Liu团队开发了多种PE6变体(PE6a-g),每种都有其特定的优势,以适应不同类型的基因编辑需求,并提供了一个决策树,帮助研究者根据特定的编辑需求选择合适的PE6变体。
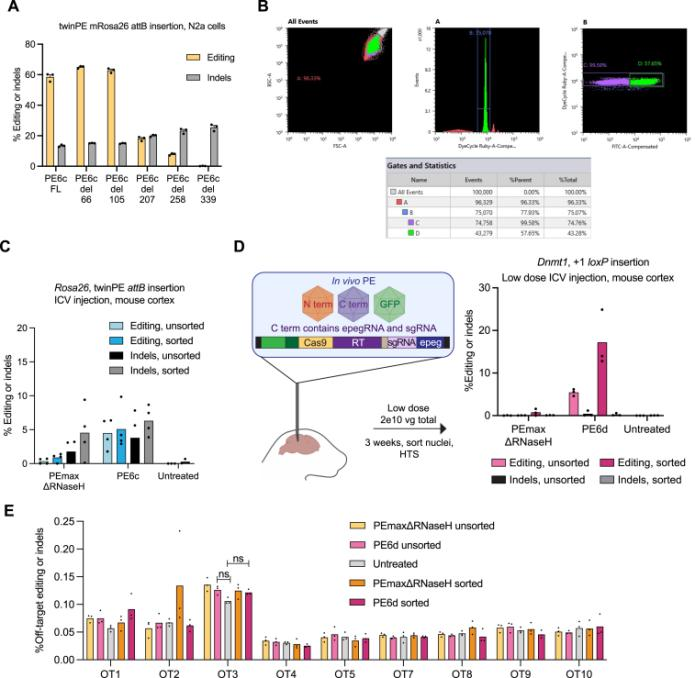
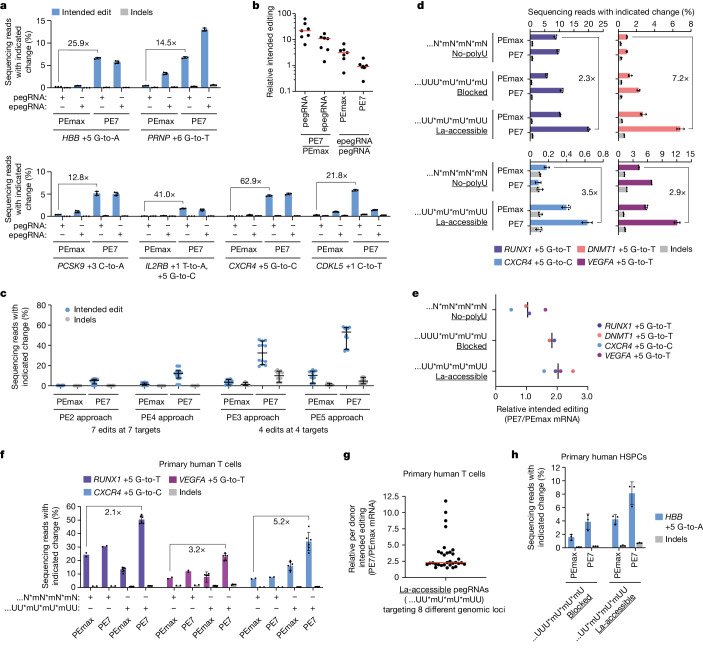
2024年4月,美国普林斯顿大学Britt Adamson和Lance R. Parsons团队在Nature上发表了名为:Improving prime editing with an endogenous small RNA-binding protein[5]的文章,发现影响PE效率的关键调控因子:La protein,并开发了一种新的PE系统——PE7。PE7在8个不同的基因组位点(如镰状细胞病相关的HBB基因、朊蛋白病相关的PRNP基因、家族性高胆固醇血症相关的PCSK9基因等)、3种细胞系(HEK293T、HeLa和U2OS)以及不同类型的编辑(单核苷酸替换、插入或15碱基对的缺失)中都显示出了效率的提高,特别是在具有完整错配修复(MMR)系统的HeLa和U2OS细胞中改进最为显著,与PEmax相比,PE7在U2OS细胞中使用pegRNA和epegRNA进行编辑时,显示出21.8倍和10.8倍的中位数提升。PE7在原代人类T细胞和CD34+造血干细胞和祖细胞(HSPCs)中进行编辑时,与La-accessible pegRNAs配对使用,相比PEmax显示出更高的目标编辑效率。研究人员还测试了带有La(1–194)结合RNA能力受损的PE7突变体,发现这些突变消除了La(1–194)融合到PEmax时的改进效果,证实了La蛋白通过与pegRNA 3'端相互作用来促进Prime editing的模型。
2024年4月29日,首个Prime Editing基因编辑疗法获批临床。David Liu所在的Prime Medicine 公司宣布,其治疗慢性肉芽肿病(CGD)的先导编辑疗法的试验性新药(IND)申请已获得美国食品药品监督管理局(FDA)的批准,将启动全球1/2期临床试验。这项1/2期临床试验将评估Prime editor疗法在成年人和儿童慢性肉芽肿病患者中的安全性、生物活性和初步疗效。最初的临床试验参与者将是病情稳定的成年人,在这些患者中证明了安全性和生物活性后,将进一步纳入发生持续感染或严重炎症的患者以及青少年和儿童患者。临床试验参与者将进行安全性随访,包括造血系统的植入和重建,恢复免疫功能的早期生物标志物,以及慢性肉芽肿病相关感染和炎症并发症的长期解决和预防。
结语
回顾基因编辑技术从ZFN到Prime Editing的跨越式发展,我们见证了生物技术在精确操控生命遗传密码方面取得的巨大进步。这些技术不仅为我们提供了深入理解生命奥秘的窗口,也为疾病治疗、作物改良等领域带来了前所未有的可能性。尽管每种技术都有其自身的优势和局限性,但正是这些技术的不断完善和创新,推动着生命科学研究的深入发展。随着生物技术的不断进步和交叉学科的融合,基因编辑技术将在更多领域展现出其强大的应用潜力,期待这些技术能够更加成熟、更加高效、更加安全,为人类的健康和福祉做出更大的贡献。
- 艾迪基因深耕基因编辑领域多年,专注CRISPR/Cas底层研发,基于Prime Editing先导编辑技术,升级研发Bingo™先导编辑基因定点突变平台,已为全球各科研院所及企业提供精准、高效的基因定点突变细胞定制服务。

近期资讯
1.【本月之星】泛素化相关文库、脂质代谢文库和小鼠全基因组敲除文库
2.【基因编辑新动向】CTLA4、MUC1和mTOR基因敲除细胞
联系我们
18102225074(微信同号)
market@edgene.cn
参考文献:
[1] Komor A C, Kim Y B, Packer M S, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-4.
[2] Anzalone A V, Randolph P B, Davis J R, et al. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785): 149-57.
[3] Chen P J, Hussmann J A, Yan J, et al. Enhanced prime editing systems by manipulating cellular determinants of editing outcomes[J]. Cell, 2021, 184(22): 5635-52.e29.
[4] Doman J L, Pandey S, Neugebauer M E, et al. Phage-assisted evolution and protein engineering yield compact, efficient prime editors[J]. Cell, 2023, 186(18): 3983-4002.e26.
[5] Yan J, Oyler-Castrillo P, Ravisankar P, et al. Improving prime editing with an endogenous small RNA-binding protein[J]. Nature, 2024, 628(8008): 639-47.



