【文献解读】利用Cas12a敲除技术探索跨物种lncRNA功能保守性

今年一月,清华大学和北京大学的研究人员在Nature Genetics上发表题为Computational prediction and experimental validation identify functionally conserved lncRNAs from zebrafish to human的研究论文,影响因子30.8。该研究使用CRISPR-Cas12a对人类细胞中预测有斑马鱼同源物的coPARSE-lncRNAs进行基因敲除以产生细胞增殖缺陷,通过功能救援实验(KO-rescue)将预测的斑马鱼同源物引入敲除人类lncRNA的细胞中尝试修复该缺陷,同时在斑马鱼胚胎中进行类似实验,证明lncRNAs在跨物种中的功能保守性,揭示lncRNAs在细胞增殖和发育中的功能,为癌症治疗领域提供新的治疗策略。

长链非编码RNA(lncRNAs)是一类长度大于200nt的长链非编码RNA,可以作为许多生理过程和疾病的调节因子其失调与多种病理过程有关,大多数lncRNA的功能仍然未知,并且由于它们的最小序列保守性,许多lncRNA可能没有功能。比较序列分析可以为剖析lncRNA的进化和功能提供有用的信息,通过序列分析可以寻找功能保守的lncRNA,然而绝大多数lncRNA显示出很少的序列相似性,lncRNA进化和蛋白质编码基因进化具有本质上的不同,因此迫切需要一种识别远缘物种lncRNA同源物的创新策略。
一、候选lncRNA同源物的同源性鉴定及coPARSE-lncRNA进化同源物的鉴定
为了探索lncRNA的同源性,研究人员对牛、负鼠、鸡、蜥蜴、青蛙和斑马鱼在内的六种脊椎动物的lncRNA数据集进行了注释,作为对GENCODE项目中现有的高质量人类和小鼠lncRNA注释的补充,发现整理的lncRNA与其他五个公共来源的lncRNA有广泛的重叠。整理的6种脊椎动物物种的20,688-42,725个候选lncRNA与蛋白质编码基因相比始终表现出较低的蛋白质编码潜力、较低的表达水平和较高的组织特异性,且序列保守性非常低,8种脊椎动物(前六种加上人和鼠)的成对BLAST分析中来自一个物种的lncRNA只有0.3-3.9%与来自另一个物种的lncRNA具有可检测的序列相似性,远低于蛋白质编码基因的水平(40-90%)。
Synteny分析可以识别出具有相同进化起源的基因组区域块,研究人员推测synteny information可能有助于鉴定保守的lncRNA。为此,研究人员设计了一个基于合成性识别人类lncRNA的候选lncRNA同源物预测模型,以蛋白质编码同源对及其相关分数作为模型的训练集,用于预测lncRNA的同源关系。该分析在其他物种中发现了数千种人类lncRNA基因的同源对应,已鉴定的合成lncRNA候选基因的基因组背景与同源蛋白质编码基因的基因组背景非常相似。
RBP是RNA的重要调节因子,研究人员推测RBP结合位点的一致模式可以为鉴定功能保守的lncRNA同源物提供信息。为此,研究人员为所研究的8个物种定义了RBP结合基序库,预测了其他脊椎动物中570-5,564种人类lncRNA的同源物,并将其定义为coPARSE-lncRNAs,其中570个人类coPARSE-lncRNAs可预测斑马鱼的同源性,表明合并保守的RBP结合位点数据大大提高了预测潜在lncRNA同源物的准确性。
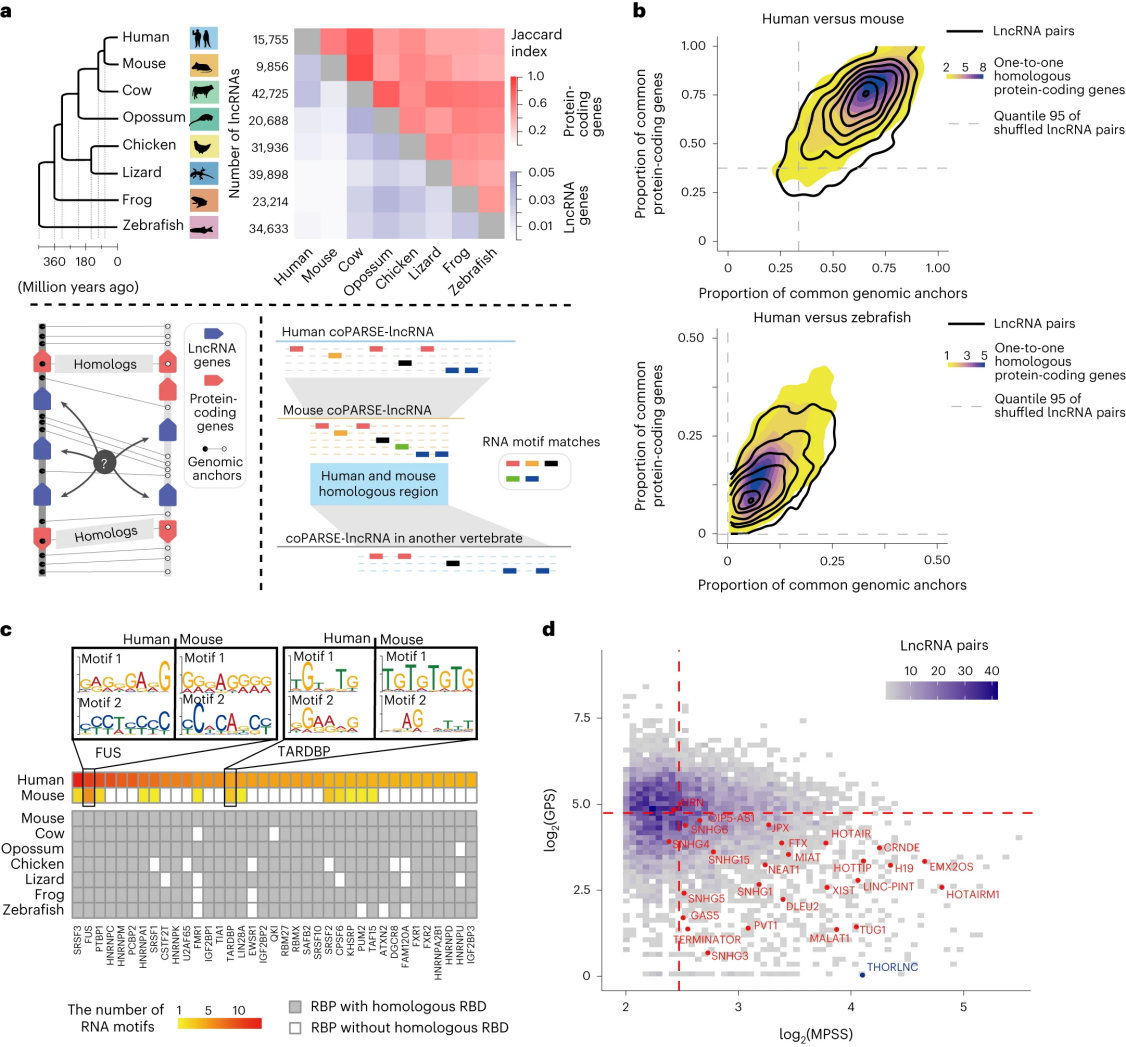
图1 在脊椎动物中鉴定coPARSE - lncRNA及其同源物
二、lncRNA同源物的进化和功能特征
研究人员将coPARSE-lncRNA及其同源物分为两组:homolog_ss组包含605对序列相似度高(>50%)的coPARSE-lncRNA同源物;以及homolog_nss组包含另外4959对序列相似度低或无序列相似度的coPARSE-lncRNA同源物。比较两组中coPARSE-lncRNA同源对的序列保守性,发现对于人类-小鼠组和人类-斑马鱼组,homolog_ss组的保守性比homolog_nss组高得多。对于homolog_ss组和homolog_nss组,基序区域的常见单核苷酸多态性(snp)密度或主要可选等位基因频率都比非基序区域低得多。这些结果表明,coPARSE-lncRNAs的预测基序区域比非基序区域经历了更大的选择压力。
相比于随机lncRNA,coPARSE-lncRNA同源物具有相对较高的组蛋白修饰模式相似性,且在不同物种中表现出可比的组织表达谱,两者都高于其他随机的同源lncRNA对。此外,通过全基因组关联研究,570个人类coPARSE-lncRNA中有270个位于与疾病相关的基因组区域高于其它人类lncRNA。相比于随机lncRNA,人类coPARSE-lncRNA富含疾病相关突变,其表达在癌症组织中更容易失调。
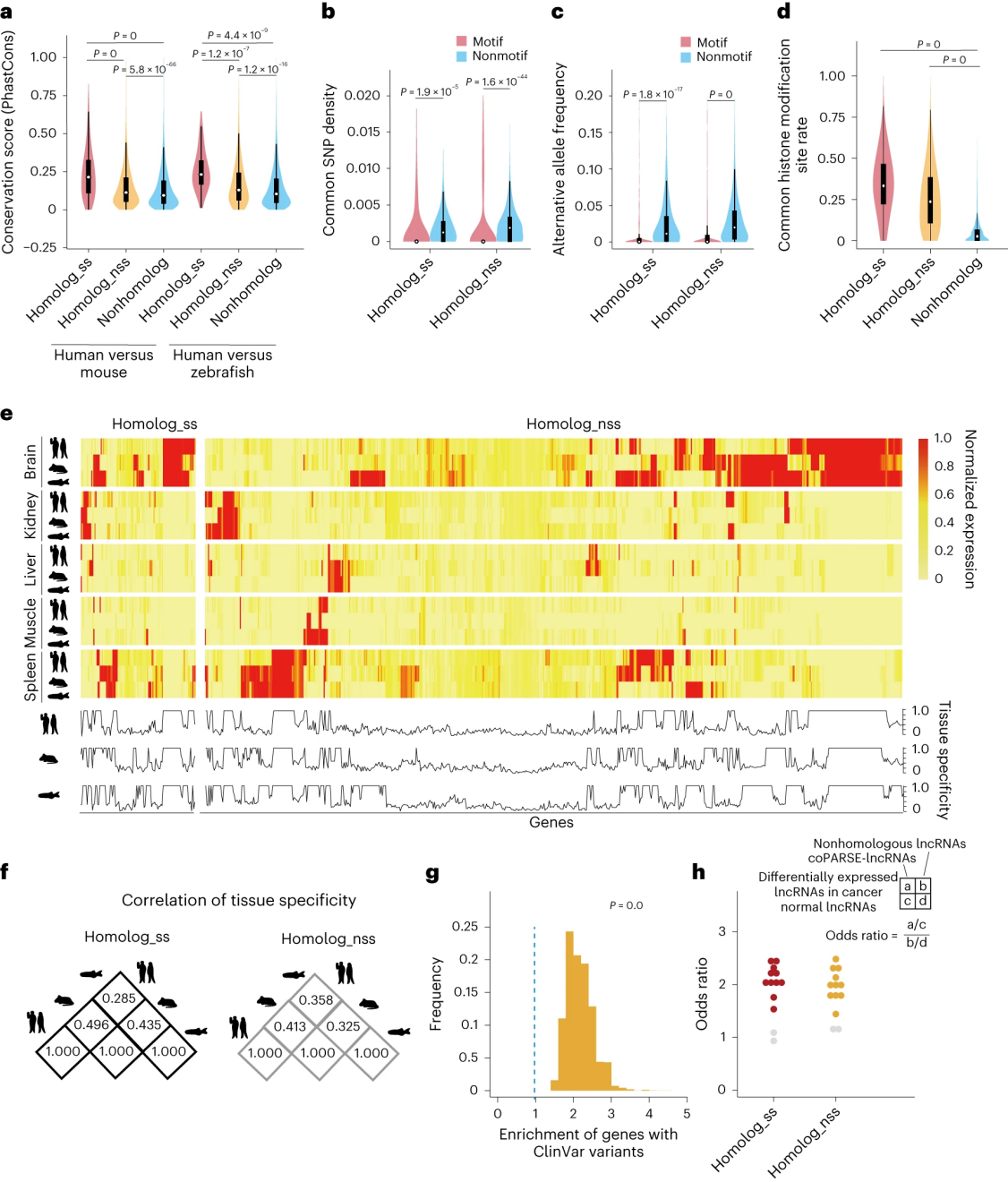
图2 coPARSE - lncRNAs及其预测的同源物具有相似的进化和功能特征
三、CRISPR筛选鉴定促进增殖的lncRNA
研究人员为了对coPARSE - lncRNAs进行功能表征,利用癌细胞系对人类癌症样本中高表达的574种人类lncRNAs(包括249种在斑马鱼中预测同源的coPARSE-lncRNAs)进行了细胞增殖试验,并为574个lncRNA各设计了20对crRNA,通过慢病毒转染将文库导入稳定表达Cas12a的三种癌细胞系(HeLa、Huh7和MCF7)中,并选择绿色荧光蛋白(GFP)阳性细胞进行繁殖,以进行广泛的CRISPR KO筛选。
研究人员在三种癌细胞系中鉴定出167种lncRNA(75种coPARSE-lncRNA),与第0天相比,它们在第15、30和45天的crRNA丰度显著降低,82%靶向这些基因的crRNAs被耗尽,不同细胞系之间存在有限的重叠。重点研究几个负向选择的coPARSE-lncRNA以验证筛选结果,对比阳性对照lncRNA RNY1和两个候选的coPARSE-lncRNA(RP1-212P9.3和AL355075.4),经Cas12a 敲除后lncRNA所在细胞的增殖率大幅降低。三个coPARSE-lncRNA(RP1-212P9.3, AL355075.4和RP11-563J2.3)的shRNA敲低证实了它们促进细胞增殖的功能,与RP1212P9.3部分重叠的蛋白编码基因OPRD1的KO不影响细胞增殖,通过CRISPR Cas12a 敲除筛选确定了调节癌细胞增殖的保守的coPARSE-lncRNA。
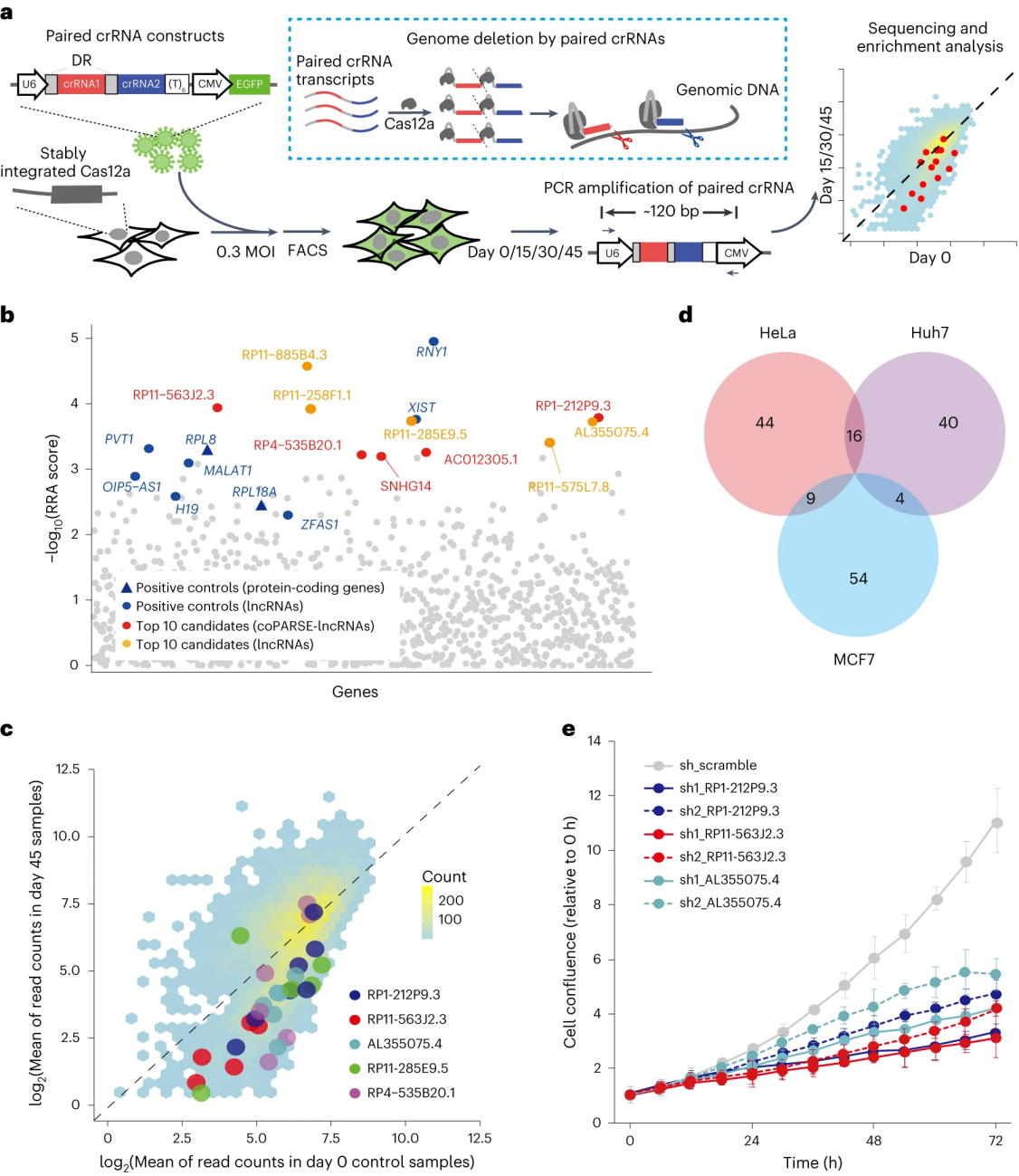
图3 CRISPR - Cas12a筛选并验证coPARSE - lncRNA的功能
四、lncRNA同源物的保护功能验证
研究人员使用CRISPR-Cas12a KO-rescue系统探索了coPARSE - lncRNA的功能保守性,在成功测试了多西环素(Dox)诱导的异位基因表达后,利用靶向21个人类lncRNA的慢病毒颗粒转染Cas12a表达的癌细胞,并使用斑马鱼同源物的拯救序列转染表达Cas12a的癌细胞。增殖实验显示,所有选择的coPARSE - lncRNA中(除RP11-20I23.6外),与对照组相比,no-Dox组的增殖率下降了30-70%。相比于no-Dox组,在含Dox培养基中生长的细胞对5个斑马鱼同源物的增值率增加,表明这5个斑马鱼同源物对促进增殖进行了功能补偿。以coPARSE - lncRNARP1-212P9.3为例,斑马鱼同源区域的异位表达部分挽救了RP1-212P9.3 KO导致的细胞增殖缺陷,而萤火虫荧光素酶基因片段长度匹配的表达则没有挽救作用。
研究人员还评估了预测的coPARSE - lncRNA同源物在斑马鱼早期胚胎发育中的潜在功能保守性。对于在拯救实验中鉴定的四个coPARSE - lncRNA(RP1-212P9.3、RP11-1055B8.4、RP11-429B14.1和RP11-223I10.1),研究人员敲除斑马鱼早期胚胎中预测的同源物,并通过形态学判断观察到明显的发育迟缓,而人类coPARSE - lncRNA同源物的正义序列挽救了发育延迟,使其正常发育。这些结果表明,coPARSE - lncRNA在远亲物种中具有共同的调控作用。
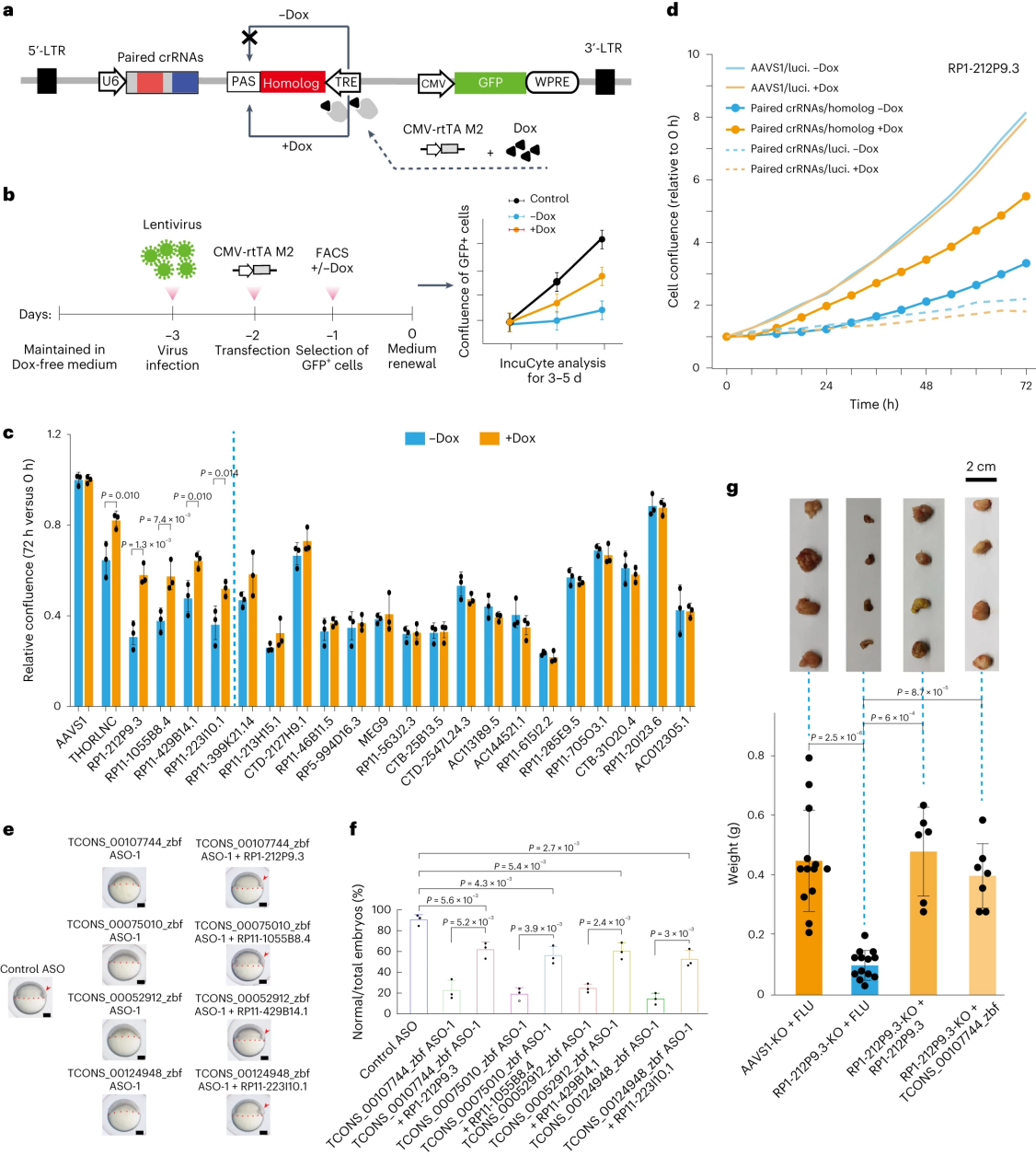
图4 coPARSE - lncRNAs的功能验证
五、coPARSE-lncRNAs同源互作组之间存在较大的重叠
为了测试coPARSE - lncRNAs同源对是否与相同的RBP相互作用,研究人员对RP1-212P9.3和RP11-1055B8.4进行了RNA下拉和质谱分析,以检测人、小鼠和斑马鱼lncRNA同源物与HeLa细胞裂解物的相互作用蛋白。下拉蛋白的主成分分析(PCA)显示,在包埋物中,相对于同一物种的不同lncRNA,coPARSE-lncRNA同源对彼此之间的结合蛋白谱更接近,这有力地支持了coPARSE-lncRNA同源对之间结合蛋白谱的相似性。免疫印迹证实,人类coPARSE-lncRNA RP1-212P9.3及其小鼠和斑马鱼同源物均与CAPRIN1、TARDBP和NONO相互作用。RNA pull-down实验从RBP文库中鉴定出6个和5个RBP,用于结合RP1-212P9.3和RP11-1055B8.4进行基序模式分析,鉴定的RP1-212P9.3的6个RBP中有3个,RP111055B8.4的5个RBP中有2个被准确预测,且同源对之间的基序匹配良好。
研究人员还对小鼠细胞和早期斑马鱼胚胎中的RP1212P9.3同源物进行了RNA pull-down和MS分析,结果显示在人类HeLa细胞、小鼠V6.5细胞和斑马鱼胚胎中,RP1212P9.3同源物的富集RBP具有很强的相关性和广泛的重叠。此外,研究人员评估了RP1-212P9.3同系物在相应物种细胞中的共同蛋白。与基准相比,RP1-212P9.3的下拉蛋白与其同源物之间存在相对较高的重叠,被RP1-212P9.3同源物拉低的蛋白显示出翻译和细胞增殖相关功能的富集。
研究人员还进行了五次额外的互补试验,使用预测的人类同源RP1-212P9.3片段,其中包含不同的假定RBP结合位点,试图挽救由TCONS_00107744_zbf敲低引起的斑马鱼早期发育延迟缺陷。结果显示,只有NONO结合位点完整的片段才能挽救TCONS_00107744_zbf敲低的斑马鱼胚胎的发育延迟,证明RBP (NONO)结合位点对救援片段的特异性。研究人员还在HeLa细胞中对coPARSE-lncRNA(RP1-212P9.3和RP11-1055B8.4)进行了两次KO-rescue实验,发现NONO结合位点和IGF2BP2结合位点的突变分别消除了TCONS_00107744_zbf片段和TCONS_00075010_zbf片段的拯救作用。结合斑马鱼救援实验的结果,表明coPARSE-lncRNA同源物与特定的RBPs相互作用在功能上是相关的。
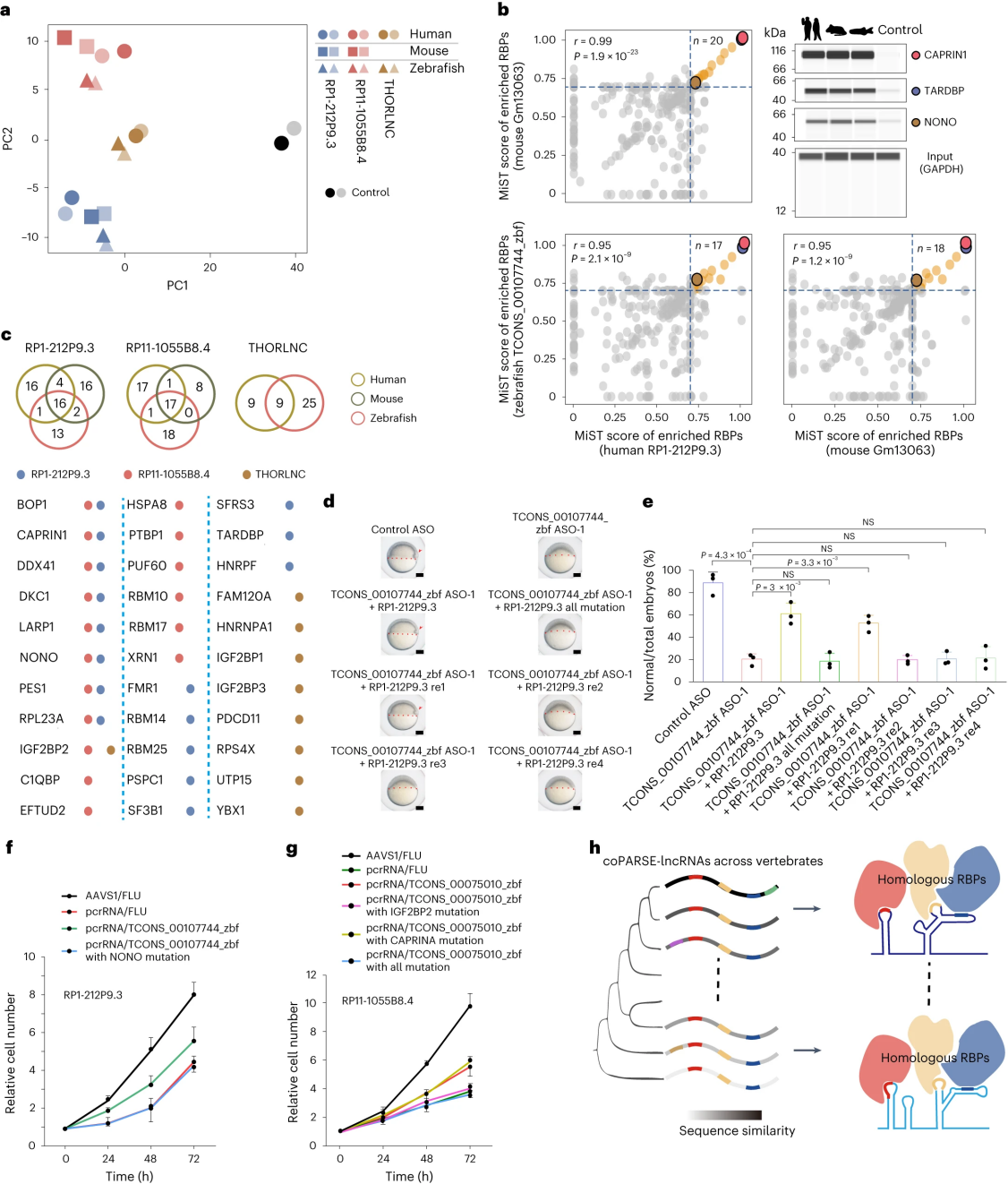
图5 2个coPARSE - lncRNA的RBP互作组鉴定及功能分析
综上所述,研究人员利用CRISPR Cas12a基因敲除技术和KO-rescue技术,将预测的斑马鱼同源物引入敲除人类lncRNA的细胞中以及向敲低斑马鱼lncRNA同源物的胚胎注射人类同源lncRNA片段,成功修复了敲除/敲低后导致的发育延迟,证明从斑马鱼到人类的lncRNAs在功能上具有保守性,为lncRNA功能的进化提供了新的思路
原文链接:https://doi.org/10.1038/s41588-023-01620-7
艾迪基因重榜推出CRISPR Editx™极速敲除试剂盒,使用特殊改造的CRISPR Cas12a和创新研发的CRISPR EDITx™Tran转染试剂,30min极速转染,不产生细胞损伤,ICE Analysis显示编辑效率约为95%,真正做到快速、无毒、高效率的基因编辑!
点击详情
近期资讯
2.【前沿资讯】基因编辑新动向——FGFR1、BRD4和BACE1基因敲除细胞
联系我们
18102225074(微信同号)
market@edgene.cn




